RNA-polymerase
RNA-polymerase of DNA-dependent RNA polymerase is een enzym actief bij de transcriptie. RNA-polymerase heeft, in tegenstelling tot DNA-polymerase, een eigen helicaseactiviteit: het ontwindt een deel van het dubbelstrengig DNA (ongeveer 17 baseparen). Het heeft in tegenstelling tot DNA-polymerase geen primer nodig, de start van de transcriptie wordt hier geregeld door de aanwezigheid van een promotorsequentie.
RNA-polymerase
| ||||
 | ||||
| Identificatie | ||||
| Alt. naam | DNA-Directed RNA Polymerase | |||
| EC-nummer | 2.7.7.6 | |||
| CAS-nummer | 9014-24-8 | |||
| Databanken | ||||
| IntEnz | IntEnz view | |||
| BRENDA | BRENDA entry | |||
| ExPASy | NiceZyme view | |||
| KEGG | KEGG entry | |||
| MetaCyc | Stofwisselingsroute | |||
| PDB-structuren | ||||
| ||||
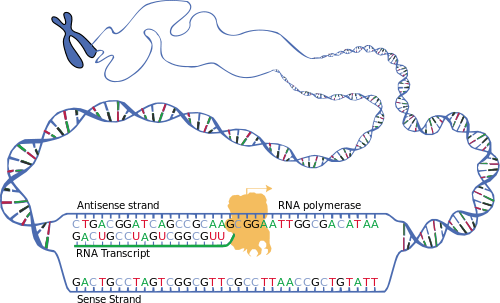
Het koppelt op een specifieke plaats (de promotor) aan een DNA- of RNA-keten en vouwt zich eromheen. Vervolgens ontwindt het het DNA of RNA en ritst als het ware de dubbelspiraal open. Daardoor wordt een stukje in de enkelstrengs-vorm tijdelijk toegankelijk voor andere actieve componenten van het enzym. Vervolgens worden steeds nieuwe nucleotiden (basen) aan de te vormen RNA-streng gekoppeld. Welk nucleotide dit zal zijn, wordt steeds bepaald door de tegenoverliggende DNA- of RNA-nucleotide. Het DNA of RNA fungeert dus als een 'mal' (men spreekt van template). RNA-polymerase beweegt zich in de zogenaamde 3' → 5'-richting over het DNA of RNA, waarbij de nieuwe RNA-keten in de 5' → 3' richting wordt gesynthetiseerd. Tegelijkertijd wordt het RNA aan de achterkant weer gescheiden van het DNA of RNA. Wanneer het hele gen gekopieerd is, koppelt RNA-polymerase los van het DNA of RNA en wordt tevens de nieuwe RNA-keten afgestoten. Bij Escherichia coli worden elke seconde ongeveer 50 nucleotiden aan de RNA-streng gekoppeld. Hierdoor wordt de RNA-streng 17 nanometer per seconde langer.
Soorten
Bij eukaryoten zijn er drie soorten RNA-polymerasen bekend met ieder een eigen functie.
- RNA-polymerase I, die betrokken is bij de productie van pre-rRNA (45S wordt omgezet in 18S; 5.8S; 28S). Daarnaast is dit enzym betrokken bij de productie van vele snRNA's (sn staat voor het Engelse small nuclear) in de celkern. Het komt voor in de celkern (nucleolus). De 14 subeenheden worden aangeduid met RPA, gevolgd door een nummer.
- RNA-polymerase II, die betrokken is bij de productie van de meeste mRNA's. Het komt voor in de celkern. De 10 - 12 subeenheden worden aangeduid met RPB, gevolgd door een nummer.
- RNA-polymerase III, die betrokken is bij de productie van tRNA, siRNA en 5S rRNA. Het komt voor in de celkern. De 17 subeenheden worden aangeduid met RPC, gevolgd door een nummer....
- RNA-polymerase IV, die betrokken is bij de productie van small interfering RNA (siRNA) in planten. De 12 subeenheden worden aangeduid met RPD, gevolgd door een nummer.
- RNA-polymerase V, die betrokken is bij de siRNA-directe de novo methylering in planten. De 12 subeenheden worden aangeduid met RPE, gevolgd door een nummer.
RNA-polymerase IV en RNA-polymerase V zijn afgeleiden van RNA-polymerase II. De 12 subeenheden worden aangeduid met RPE, gevolgd door een nummer.
Voor het scheiden van de enzymen wordt gebruikgemaakt van DEAE-Sephadex (diethylaminoethylcellulose; een ionenwisselaarhars) bij de ionenuitwisselingschromatografie. Deze techniek scheidt ze van elkaar in de volgorde van de extractie Ι, ΙΙ en ΙΙΙ door de concentratie van ammoniumsulfaat te verhogen. De enzymen werden vernoemd naar de volgorde van extractie RNA-polymerase I, RNA-polymerase II en RNA-polymerase III.[1]
Samenstelling
De RNA-polymerasen zijn zeer complex. Bij gist zijn er verschillende polypeptide-ketens met een molecuulmassa, die tussen de 7700 en 140.000 atomaire massa-eenheden ligt, bij betrokken. Verder zijn ook magnesium, zink en twee DNA-ketens er bij betrokken. Deze gist-RNA-polymerase bestaat uit meer dan 28.000 atomen.
Foutenherkenning
RNA-polymerasen beschikken over een eenvoudig mechanisme voor foutenherkenning. Wanneer zich aan een DNA-streng een verkeerd RNA-nucleotide bindt, blijft RNA-polymerase langer op de betreffende DNA-plek stilstaan. Hierdoor wordt de kans groter dat het verkeerde RNA-nucleotide weer loslaat van het DNA en is de foutenkans 1 op 10.000 basen. Dit komt overeen met ongeveer 1 fout per gesynthetiseerd RNA-molecuul.
Bepaling type RNA-polymerase
Alfa-amanitine wordt door zijn werkingsmechanisme ook veelal gebruikt als een ‘tool’ in wetenschappelijke studies in moleculaire biologie en biologisch onderzoek. Het kan gebruikt worden om te bepalen welke vormen van RNA-polymerase aanwezig zijn. Men test dan de gevoeligheid van de RNA-polymerase in aanwezigheid van alfa-amanitine. RNA-polymerase I is ongevoelig, RNA-polymerase II zeer gevoelig en RNA-polymerase III is enigszins gevoelig voor alfa-amanitine.[2]
RNA-polymerase-transcriptiecyclus
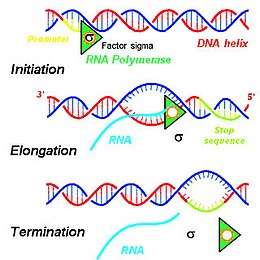
Bij de transcriptie worden drie hoofdstadia onderscheiden:
- Initiatie (aanzetten): de binding van het RNA-polymerasecomplex aan de promotoren van de genen met behulp van transcriptiefactoren.
- Elongatie (verlengen): de actuele transcriptie van het merendeel van genen in de overeenkomstige RNA-sequentie.
- Terminatie (beëindigen): het stoppen van de RNA-transcriptie en het ontmantelen van het RNA-polymerasecomplex.
Initiatie
Bij bacteriën is bij de binding van RNA-polymerase de sigmafactor betrokken voor het herkennen van de core-promotorplaats dat de -35- en -10-elementen bevat (liggend voor het begin van de te transcribeerde sequentie) en ook, bij sommige promotoren, het α-subeenheid-C-terminaal-eiwitmotief, dat de promotor bovenstroomse elementen herkent. Dit zijn meervoudig verwisselbare sigmafactoren, die ieder een bepaalde set van promotoren herkend. Bijvoorbeeld in Escherichia coli komt onder normale condities σ70 tot expressie en herkent dit de promotoren voor de betrokken genen ("huishoudgenen"), terwijl σ32 promotoren genen herkent, die bij hoge temperaturen tot expressie komen ("hitte-schokgenen").
Nadat RNA-polymerase aan het DNA is gebonden, gaat RNA-polymerase van een gesloten over in een open vorm. Hierdoor gaan de DNA-strengen gedeeltelijk, ongeveer 13 basenparen, uit elkaar. Ribonucleotiden binden zich aan de DNA-streng door de Watson-Crick basenpaarbindingsinteracties. Omdat de DNA-plekken voor RNA-polymerase ontwonden zijn, moet er op een andere plaats een sterkere winding (supercoiling) optreden. Plekken achter de RNA-polymerase hebben een sterkere binding in tegengestelde richting.
Het contact van RNA-polymerase met de promotorplaats beperkt de mogelijkheid van het enzym om contact te maken met het DNA benedenstrooms. Daarom maakt het RNA-polymerase als de open vorm gestabiliseerd is een RNA-streng op het actieve centrum voor een DNA-RNA-heteroduplex (~ 8-9 basenparen), waardoor het elongatiecomplex wordt gestabiliseerd.
Voor het afmaken van de RNA-streng moet RNA-polymerase contact blijven houden met de promotorplaats wanneer het DNA verder benedenstrooms wordt ontwonden. Tijdens de overgang naar het loslaten van de promotor komt RNA-polymerase onder spanning. Op het moment dat de DNA-RNA-heteroduplex lang genoeg is, komt RNA-polymerase los van de bovenstroomse contacten en gaat RNA-polymerase over in de elongatiefase.
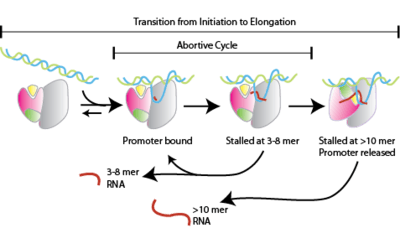
Bij het gepauzeerde transcriptiecomplex zijn er twee mogelijkheden:
- Het stoppen van het transcript in wording en beginnen met een nieuwe transcriptie op de promotorplaats. Dit wordt de gearborteerde transcriptie genoemd. Er wordt een 3-8 basenpaar lange RNA-streng gevormd.
- Een nieuwe 3'-OH-verbinding maken op de actieve plaats van het in wording zijnde transcript door katalytische activiteit en het DNA verder transcriberen zonder dat RNA-polymerase beweegt, maar door het DNA naar zich toe te trekken (scrunging). Er wordt een meer dan 10 basenpaar lange RNA-streng gemaakt.
Elongatie
Bij de verdere transcriptie gaat het open complex over in het transcriptiecomplex. Op het moment dat RNA-polymerase begint met het maken van langere RNA-strengen komt het los van de promotor, wordt het contact met de -10- en -35-elementen verbroken en houdt bij bacteriën de σ-factor de RNA-polymerase op zijn plaats. Er wordt een nieuwe 3'-OH-verbinding gemaakt op de actieve plaats van het in wording zijnde transcript door katalytische activiteit.
Aspartyl-resten (asp) van RNA-polymerase binden aan de Mg2+-ionen, die op hun beurt de fosfaten van de ribonucleotiden coördineren. Het eerste Mg2+-ion bindt aan het α-fosfaat van het toe te voegen nucleosidetrifosfaat (NTP). Hierdoor wordt de nucleofiele binding met het 3'-OH van het RNA-transcript mogelijk en wordt opnieuw een nucleosidetrifosfaat aan de RNA-streng toegevoegd. Het tweede Mg2+-ion bindt aan het pyrofosfaat van het nucleosidetrifosfaat. De nettoreactievergelijking is:
(NMP)n + NTP --> (NMP)n+1 + PPi (NMP = nucleosidemonofosfaat, NTP = nucleosidetrifosfaat en PPi = het anion-deeltje van pyrofosfaat)
Reactie bij mRNA
De verlenging van het mRNA door de transcriptie van de gekoppelde DNA-enkelstreng wordt gekatalyseerd door de RNA-polymerase II. In dit voorbeeld een ribulose-molecuul, dat al deel uitmaakt van het mRNA en de uracil-nucleotide UTP (uridinetrifosfaat):
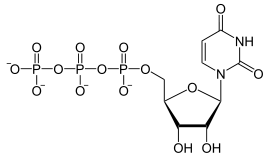

De fosfaatgroep van het UTP wordt met de hydroxylgroep van het ribulose van het mRNA verestert. Het RNA-molecuul wordt verlengd onder afsplitsing van pyrofosfaat (PPi).
Terminatie
RNA-polymerase III stopt de transcriptie op een klein polyT's (polythymines)-gedeelte (5-6 basenparen). Bij mRNA gemaakt door RNA-polymerase II vindt na de terminatie polyadenylatie plaats.
Prokaryoten
In prokaryoten is de terminatie van de RNA-transcriptie rho-onafhankelijk of rho-afhankelijk.
De rhofactor (ρ-factor) is een eiwit dat bindt aan de transcriptie-terminator-pauzeplaats. Deze plaats is een enkelstrengs RNA, dat 72 nucleotiden lang is en achter de open leesraam ligt. Het is een C-rijke/G-arme (cytosine-rijke/guanine-arme) sequentie zonder secundaire structuur.[3]
Bij de rho-onafhankelijke transcriptie-terminatie stopt de transcriptie zonder hulp van het rho-eiwit. Transcriptie van een palindromische DNA-regio zorgt voor een haarspeldstructuur van de RNA-transcriptie lus en bindt aan zichzelf. De haarspeldstructuur is vaak rijk aan G-C-basenparen (bp), waardoor het stabieler is dan de DNA-RNA-hybride. Het resultaat is dat de DNA-RNA-hybride van 8 bp van het transcriptiecomplex overgaat in een 4bp-hybride. Deze 4bp-hybride heeft een zwakke A-U (adenine-uracil)-basenparing, waardoor het volledige RNA-transcript los laat van het DNA.
Hogere eukaryoten
Bij hogere eukaryoten bindt en buigt de transcriptieterminatiefactor RNA-polymerase I (TTF-I) de terminatieplaats aan het 3'-eind van het getranscribeerde gebied. Hierdoor wordt RNA-polymerase I gedwongen te pauseren. TTF-I zorgt er met de hulp van de polymerase-I en transcriptie-releasefactor (PTRF) en een T-rijk gebied voor, dat RNA-polymerase I de transcriptie staakt en loslaat van het DNA en de nieuw getranscribeerde sequentie.
Externe link
Zie ook
Bronnen, noten en/of referenties
|