Pyrrolysine
Pyrrolysine (in de eiwitchemie afgekort als Pyl of O) is een natuurlijk voorkomend, en tijdens de translatie van de genetische code direct in het groeiend eiwit ingebouwd aminozuur. PYL wordt door een aantal methaanproducerende archaea en, voor zover bekend (2015), één bacterie gebruikt in enzymen die deel uitmaken van het methaanproducerende metabolisme. Het aminozuur is een derivaat van lysine, maar dit is via een peptidebinding aan de aminogroep van de zijketen gekoppeld aan een pyrrolinering. De genetische code voor dit aminozuur in de betreffende organismen wijkt af van de standaardwijze waarop aminozuren gecodeerd worden. Omdat PYL tijdens de translatie in de groeiende peptideketen wordt ingebouwd, wordt het beschouwd als het 22e (na selenocysteïne, het 21e) proteïnogene aminozuur.[1][2][3]
Pyrrolysine
| ||||
| Structuurformule en molecuulmodel | ||||
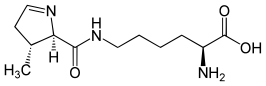 | ||||
| Algemeen | ||||
| Molecuulformule (uitleg) | C12H21N3O3 | |||
| IUPAC-naam | N6-{[(2R,3R)-3-methyl-3,4-dihydro-2H-pyrrol-2-yl]carbonyl}-L-lysine | |||
| Molmassa | 255,313 g/mol | |||
| SMILES | O=C(NCCCC[C@@H](C(=O)O)N)[C@@H]1/N=C\C[C@H]1C | |||
| CAS-nummer | 448235-52-7 | |||
| PubChem | 5460671 | |||
| ||||
De gezamenlijke nomenclatuurcommissie van de IUPAC/IUBMB heeft een officiële aanbeveling gedaan de drieletterafkorting Pyl te grbuiken Voor de éénlettercodering van aminozuren wordt de O aanbevolen.[4]
Samenstelling
Uit Röntgendiffractie[5] en MALDI massaspectrometrie blijkt dat PYL is opgebouwd uit 4-methylpyrroline-5-carbonzuur dat via een peptidebinding gekoppeld is aan de aminogroep in de zijketen van lysine.[6]
Biosynthese
De biosynthese van PYL gaat uit van twee moleculen L-lysine. De eerste stap wordt gevormd door de omzetting van een van de lysinemoleculen in (3R)-3-Methyl-D-ornithine, dat vervolgens aan het tweede lysinemolecuul gekoppeld wordt. Vervolgens wordt een NH2-groep geëlimineerd, gevolgd door de cyclisatiereactie en de dehydrogenering waarbij ten slotte L-PYL ontstaat.[7]
Katalytische werking
De volgende beschrijving is een mogelijk, maar nog niet (2015) bewezen, reactiemechanisme.
De extra pyrrolinering van PYL komt voor in de actieve plaats van verschillende methyltransferases. Aangenomen wordt dat de ring relatief vrij kan roteren. Daardoor kan deze ring gebruikt worden om de methylgroep van methylamine op de juiste wijze te positioneren en te activeren voor de reactie met een corrinoide-cofactor. Een in de buurt liggende carboxylaatgroep (van glutamine) wordt geprotoneerd, waarna dit proton overgedragen wordt op het stikstofatoom van het stikstofatoom in de ring, waardoor methylamine een nucleofiele additiereactie op het ringkoolstof kan uitvoeren. De positieve lading die op het stikstofatoom in de ring is ontstaan wordt door de negatieve lading van het glutamaat in de juiste positie ten opzichte van de actieve plaats gebracht, waardoor de methylgroep van het methylamine kan reageren met het kobaltatoom in het corrinoide. Op deze manier wordt netto een CH3+-groep overgebracht naar het kobalt-atoom met een gelijktijdige verandering van oxidatiegetal van I naar III. Onder uitstoting van NH3 wordt het oorspronkelijke imine hersteld.[5]
De genetische code voor pyrrolysine
Algemeen
Een van de voornaamste functies van de genetische code is het aansturen van de opbouw van proteïnen en wanneer en in welke cel(onderdeel) de synthese plaatsvindt. Bijna alle proteïnen worden opgebouwd uit slechts 20 standaard bouwstenen, aminozuren, die op basis van de in DNA vastliggende genetische code in een lange keten aaneen gekoppeld worden. Voor speciale chemische reacties wordt vaak na de translatie nog een posttranslationele modificatie uitgevoerd. Toch is de genetische code zelf vrijwel voor alle organismen identiek. Onderzoekers kunnen daarom op basis van de volgorde van DNA van een nieuw organisme direct aangeven wat de waarschijnlijke functie ervan is, gebaseerd op die universele code. Voorwaarde is dan wel, dat de code echt universeel is. De ontdekking van ongebruikelijke aminozuren, gecodeerd met een eigen code, zet vraagtekens bij de universaliteit van de genetische code. Het is dan belangrijk te begrijpen, wat er precies aan de hand is. Daarnaast vormen dit soort afwijkingen een aanwijzing, dat de evolutie van de genetische code niet gestopt is voor de laatste gemeenschappelijke voorouder van alle rijken in de biologie, ongeveer drie tot vier miljard jaar geleden.
De twee tot nu toe (2015) ontdekte ongebruikelijke gecodeerde aminozuren zijn selenocysteïne en PYL. PYL is in 2002 ontdekt als onderdeel van de actieve plaats van het methyl-transferase van het methaanproducerende archeon Methanosarcina barkeri.[5][8] De genetische code voor PYL is UAG, normaal gesproken een stopcodon. De synthese en de inbouw van PYL in proteïnes wordt aangestuurd door het genencluster pylTSBCD.[9]
Codering
In tegenstelling tot een aantal andere lysinederivaten die tijdens een posttranslationele modificatie gerealiseerd worden (bijvoorbeeld: hydroxylysine, methyllysine en hypusine), wordt PYL tijdens de translatie als zodanig in de eiwitketen ingebouwd, vergelijkbaar met de standaard aminozuren. In het mRNA wordt het triplet UAG gebruikt, dat voor de meeste organismen een stopcodon vormt. Het UAG-triplet gebruiken als code voor PYL vraagt slechts de aanwezigheid in het genoom van twee genen: het pylT-gen, dat codeert voor het tRNA met een CUA-anticodon , en het pylS-gen, dat codeert voor het enzym dat het tRNA laadt met PYL. Het UAG-codon wordt gevolgd door een PYLIS downstream sequence.[10] Deze speciale manier van coderen biedt bovendien de mogelijkheid om ten behoeve van researchdoeleinden ook andere aminozuren via deze route in eiwitten in te bouwen. Onder andere in Escherichia coli zijn hier reeds resultaten mee geboekt.[11][12][13][14]
Evolutie
De genen voor pylT (tRNA) en pylS (koppelenzym) komen in Methanosarcina barkeri in hetzelfde operon voor. De andere genen in het PYL-operon coderen voor enzymen die betrokken zijn bij de biosynthese van PYL. Het complete operon kan hiermee omschreven worden als een "natuurlijke uitbreidingsset voor de genetische code"[15] (Zie ook uitgebreide genetische code). Homologen komen voor in andere leden van de Methanosarcinaceaefamilie: M. acetivorans, M. mazei, and M. thermophila en een gram-positieve bacterie, Desulfitobacterium hafniense.[16][17] Bekende enzymen waarin PYL voorkomt zijn nu (2015): monomethylamine methyltransferase (mtmB), dimethylamine methyltransferase (mtbB) en trimethylamine methyltransferase (mttB). Vooral het voorkomen van het operon in Desulfitobacterium is interessant, omdat bacteria and archaea tot verschillende domeinen in de biologie behoren. Als het gebruik van PYL beperkt was tot de Methanosarcinaceae, dan kan het toepassen van dit aminozuur als een late "uitvinding" van de archaea verstaan worden.[18] Het voorkomen bij bacteriën kan op twee manieren verklaard worden:
- Het PylRS-operon was al aanwezig in de laatste gemeenschappelijke voorouder van beide domeinen, zo'n 3 miljard jaar geleden. Het operon is slechts blijven bestaan in organismen die methylamines als energiebron gebruiken.[19]
- De tweede mogelijkheid is horizontale genoverdracht, tussen twee niet gerelateerde microorganismen.[20]
Tussen de operonen van de archae en de bacteriën zijn kleine verschillen. In D. hafniense betsaat het pylS (het koppelingsenzym) uit twee eiwitten. Bovendien is het UAG-codon voor bijna alle proteïnen van dit organisme een stop-codon, met slechts één geval waarin PYL wordt gecodeerd. In tegenstelling hiermee kon bij de archae niet aangetoond worden dat UAG als stop-codon optrad.[16] Omdat er slechts een positie is waarop PYL in proteïnen wordt ingebouwd was het niet mogelijk vast te stellen of er, net als bij selenocysteïne, een soort vervolgcode noodzakelijk is om inderdaad PYL in te bouwen.
Het tRNA met CUA als anticodon kan in vitro met lysine gekoppeld worden door Lysyl-tRNA synthetases uit M. barkeri Klasse I en Klasse II. Deze enzymen werken niet met PYL. De koppeling van lysine aan het tRNA, gevolgd door aanbouw van de pyrroline-ring, is ooit voorgesteld als eerste stap in de vorming van tRNA-PYL, een reactieroute die vergelijkbaar is die voor de opbouw van tRNA-Selenocyteïne. Latere onderzoeksresultaten geven aan dat PYL direct aan het tRNA gekoppeld wordt onder invloed van de producten van het pylS-gen.[21][22] De in vitro koppeling maakt het wel mogelijk om andere, niet natuurlijke (!), aminozuren in te bouwen in proteïnen.
Bronnen, noten en/of referenties
|